Implicazioni per la biotecnologia e lo sviluppo di nuovi farmaci
Un lavoro di ricerca congiunto che ha utilizzato le simulazioni al computer, esperimenti di biochimica, e la cristallografia a raggi X di proteine per rivelare informazioni atomiche sul meccanismo dello “splicing genico”: questo è l’ultimo risultato dei ricercatori dell’IIT di Genova, guidati da Marco De Vivo, in collaborazione con il gruppo di Marco Marcia all’EMBL di Grenoble. I ricercatori hanno ottenuto istantanee dettagliate del processo di “splicing” (ovvero montaggio) in sistemi noti come introni del gruppo II, una parte specifica dell’RNA. I risultati sono stati pubblicati su Nature Communications (https://www.nature.com/articles/s41467-020-16741-4) e potranno avere implicazioni per la biotecnologia e lo sviluppo di nuovi farmaci.
“Questi risultati provengono da anni di intenso lavoro trascorso a integrare simulazioni molecolari avanzate insieme a lavori sperimentali, con l’obiettivo di spiegare questo processo biologico fondamentale regolato da un enzima RNA“, ci racconta Marco De Vivo, Responsabile del Molecular Modeling e Drug Discovery Lab dell’IIT. Questo tipo di studi sono importanti per la progettazione di nuovi farmaci e terapie genetiche specifiche, poiché consentono di rivelare nuovi meccanismi molecolari utili a risolvere specifiche malattie genetiche o infettive. “E’ stato un grande lavoro di squadra dei nostri laboratori, presso EMBL e IIT, e continueremo a collaborare su questa entusiasmante linea di ricerca“, osserva Marco Marcia, a capo del suo gruppo all’EMBL.
Lo studio di ricerca ha coinvolto due giovani ricercatori, Jacopo Manigrasso, dottorando nel laboratorio di De Vivo, e Isabel Chillón, ricercatrice Postdoc nel gruppo di Marcia, i quali sono i primi autori dello studio, avendo lavorato insieme a questa scoperta, come raccontato sul sito EMBL News (https://www.embl.org/news/science/genetic-cut-and-paste-at-atomic-resolution/).
Le simulazioni al computer sono state utilizzate per riprodurre su scala atomica il processo di “splicing” effettuato da uno specifico introne del gruppo II, una macchina molecolare analoga a ciò che nell’uomo è chiamata “spliceosoma”, cioè una complessa combinazione di molecole di RNA supportate da proteine. “Questi introni auto-splicing, chiamati introni del gruppo II, sono gli antenati evolutivi delle unità RNA dello spliceosoma, e le due macchine molecolari usano la stessa reazione chimica per eseguire lo splicing“, spiega Jacopo Manigrasso. “Comprendendo il meccanismo di auto-splicing degli introni del gruppo II, possiamo capire meglio come funziona lo splicing anche nell’uomo.”
Lo “splicing” è un passaggio cruciale nell’uso delle informazioni genetiche memorizzate nel DNA per produrre proteine, che svolgono la maggior parte delle funzioni biologiche. Per rendere utilizzabili le informazioni genetiche, il DNA deve essere convertito o trascritto in RNA messaggero (mRNA), una molecola che agisce come vettore transitorio di informazioni. Negli animali, la maggior parte dei geni e le loro trascrizioni di mRNA contengono lunghe sequenze di informazioni genetiche che non vengono utilizzate direttamente per fare parte di una proteina. Questi segmenti, chiamati introni, devono essere tagliati dal loro filamento di mRNA prima di poter essere utilizzati per produrre una proteina funzionale. Tagliando diverse combinazioni di introni da un mRNA, le cellule possono anche produrre diverse varianti proteiche da un singolo gene. Ciò consente agli organismi di aumentare il numero di proteine che possono produrre, senza dover trasportare un gene separato per ogni proteina, mantenendo il genoma compatto.
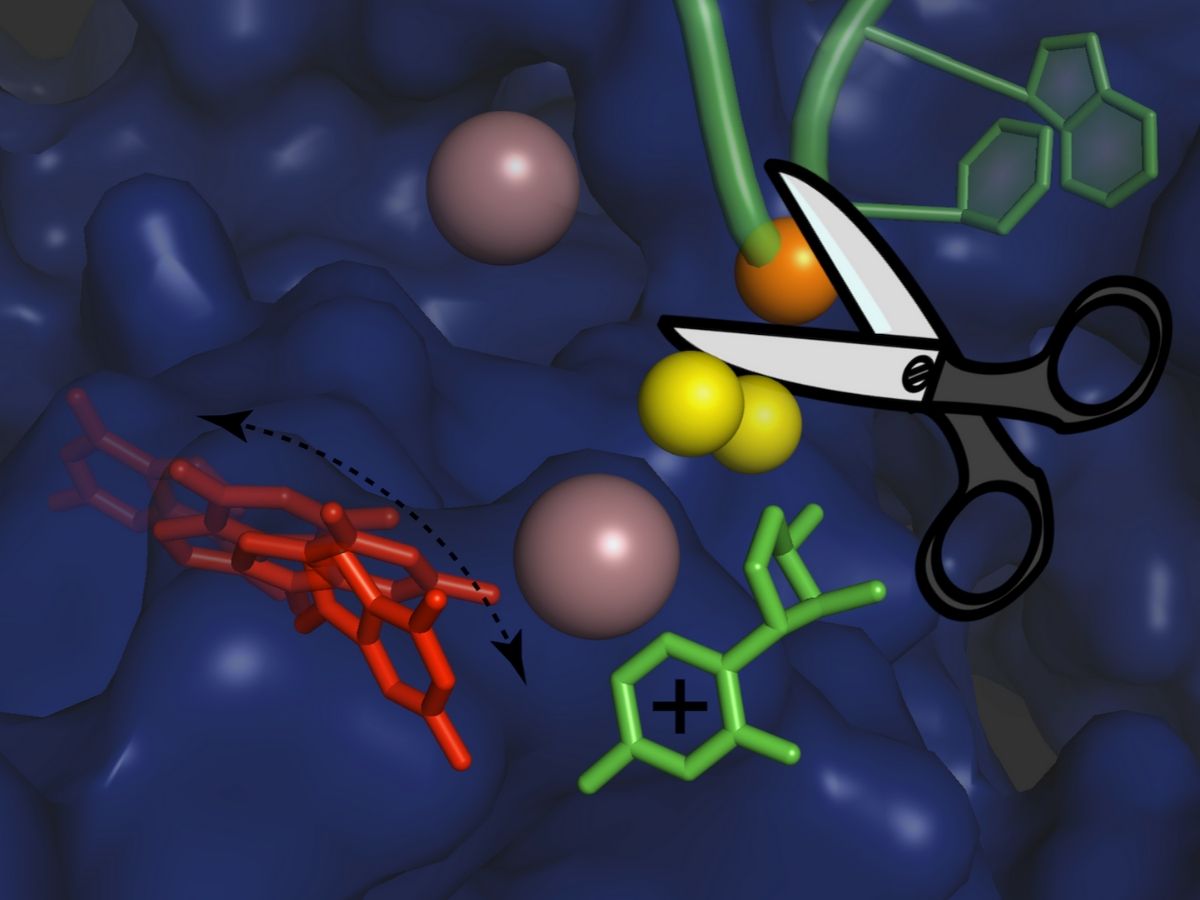
I ricercatori hanno utilizzato tecniche di cristallografia e di simulazioni al computer per capire i dettagli atomici della reazione di splicing degli introni del gruppo II. “Ogni evento di splicing consiste in due fasi principali: un taglio a un’estremità di un introne, seguito da un taglio all’altra estremità che rilascia l’introne e ricollega simultaneamente i due esoni fiancheggianti dell’mRNA“, spiega Isabel Chillón. “Durante questi passaggi l’introne deve essere leggermente riorganizzato, ma il meccanismo preciso di questa riorganizzazione non era noto fino ad ora.”
I ricercatori hanno ora scoperto proprio la chimica atomica e la fisica che definisce tale meccanismo di riorganizzazione: il primo passo della reazione di splicing avviene con il rilascio di una carica positiva all’interno dell’introne. Ciò porta al distacco temporaneo di uno ione potassio, il quale aiuta a unire e stabilizzare la struttura dell’introne, e modifica la conformazione dell’introne in prossimità del sito di taglio. Questa riorganizzazione è transitoria e permette di stabilire una configurazione adatta affinché l’introne esegua la seconda fase della reazione di splicing. Quando gli scienziati hanno impedito il trasferimento della carica positiva nei loro esperimenti, hanno scoperto che la sequenza di riorganizzazioni non ha luogo e che la seconda fase “taglia e incolla” della reazione di giunzione avviene a una velocità lenta.
Le applicazioni future di questi risultati saranno nello sviluppo di piccole molecole, chiamate inibitori dello splicing, che bloccano selettivamente gli introni di gruppo II senza influenzare lo spliceosoma. Poiché gli introni auto-splicing sono essenziali per la sopravvivenza di alcuni agenti patogeni, tali inibitori potrebbero in futuro essere utilizzati per il trattamento di malattie infettive, ampliando il nostro kit di antibiotici.